Mónica Luna | Madrid
Desde hace más de un siglo se sabe que la luz ejerce presión sobre el objeto que ilumina. Es una presión muy débil, pero de magnitud suficiente como para poder impulsar parcialmente una pequeña nave espacial mediante una gran vela solar o para poder retener o mover bolitas microscópicas mediante la utilización de haz de luz laser.
El instrumento capaz de aprovechar la 'fuerza' de la luz para atrapar objetos microscópicos se denomina 'pinzas ópticas' y gracias a él se están realizando importantes avances en el campo de la biología. Ricardo Arias-González, investigador de IMDEA
Nanociencia y del CNB-CSIC, fue pionero en introducir las pinzas ópticas en biología en España.
Con el instrumento que él mismo ha fabricado es capaz de atrapar una sola molécula para su estudio individual. Entre otros temas, ha investigado las propiedades elásticas de una sola molécula de ADN o descifrado parte del funcionamiento de los más eficientes motores nanotecnológicos que existen a día de hoy: los motores biomoleculares, responsables de la locomoción y la acción mecánica dentro de la célula.
Ricardo Arias-González junto a las pinzas ópticas. | M. L.
Mónica Luna.- ¿Cómo se pueden atrapar objetos microscópicos utilizando sólo luz?
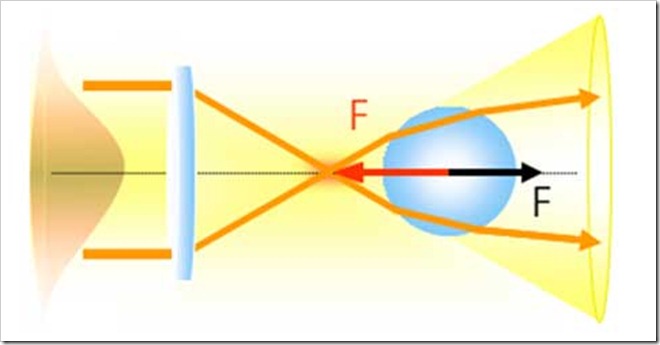
Ricardo Arias-González.- La luz ejerce dos fuerzas distintas y opuestas sobre un objeto cerca de su punto focal. Por una parte ejerce presión sobre él, lo empuja en el sentido de la propagación de la luz (flecha negra). Por otra parte, lo atrae en dirección opuesta (flecha roja) debido a los campos eléctricos que se generan. La combinación de ambas fuerzas iguales y opuestas resulta en el aprisionamiento del objeto.
Una nanopartícula atrapada mediante luz. | R. Arias-González.
M. L.- ¿Y cómo utiliza este fenómeno para estudiar las propiedades de una sola molécula?
R. A-G.- Podemos unir químicamente la molécula a dos microesferas. Una de ellas se sujeta mediante la trampa óptica y la otra con una micropipeta, como se muestra en la imagen inferior. Aumentando la distancia entre las esferas conseguimos estirar la molécula y medir sus propiedades elásticas.
En este video se puede observar cómo se consigue atrapar la bacteria 'E. coli' con las pinzas ópticas.
M. L.- ¿Cuál es la ventaja de poder estudiar las moléculas una a una?
R. A-G.- Cuando se estudian las moléculas con los métodos tradicionales, es decir introduciendo muchísimas de ellas (1023) en un tubo de ensayo, lo que analizas es el comportamiento promedio. Por ejemplo, obtendríamos una información semejante en un proceso de fabricación de automóviles del cual sólo pudiéramos conocer el comienzo, es decir, las piezas sueltas que formarán el coche y el resultado final: el coche ya terminado. Un estudio individualizado de cada molécula sería el equivalente de poder seguir el proceso de fabricación de un coche particular, podríamos saber en detalle, para ese coche concreto, cómo funcionan las máquinas que montan cada pieza y cuanto tiempo tardan en montar cada una.
M. L.- Por lo tanto, su objetivo es estudiar las moléculas de una en una para conocer sus particularidades y cómo ejecutan sus tareas.
Una sola molécula de ADN entre dos microesferas. | R. Arias-González y S. Hormeño.
R. A-G.- Exactamente. Los procesos que se dan dentro de la célula, que es la fábrica de la vida, son altamente específicos, precisos y se encuentran muy bien coordinados. Nos interesa conocer en detalle cada uno de ellos. Por ejemplo, las moléculas que se encargan de la locomoción y de las acciones mecánicas, los motores biomoleculares, no funcionan con las leyes deterministas de la física a las que estamos acostumbrados. Se rigen por la dinámica estocástica, es decir, el resultado de una acción no es siempre el mismo, por el contrario, existen distintos resultados, cada uno con una cierta probabilidad de que suceda. Desde el punto de vista de la nanotecnología es muy interesante estudiar estos motores porque el ser humano aún no ha sido capaz de fabricar algo tan eficiente. La sensación que tenemos los científicos ante los motores biomoleculares es la de estar ante una maquinaria demasiado avanzada para nuestra época. La situación sería parecida a la que experimentaría una persona del Medievo frente a un ordenador: sentiría fascinación por comprender una tecnología tan sofisticada.
M. L.- ¿Qué aspecto de esta 'ultra avanzada nanotecnología de la naturaleza' están investigando en la actualidad?
R. A-G.- Estamos interesados en conocer por qué la naturaleza ha elegido una forma determinada de almacenar la información en detrimento de otras. De la misma forma que un ordenador almacena la información en un disco duro y tiene toda una maquinaria y electrónica dedicada a leerla y copiarla, la célula también dispone de un lugar para almacenar la información: la molécula de ADN. Es un 'disco duro' con forma de hilo de doble hebra, muy fino, de 2 nm de grosor, que puede llegar a tener metros de longitud. La cadena doble del ADN es un mecanismo de seguridad: hace que los 'bits biológicos' estén más protegidos y sea más difícil perder la información, evitando enfermedades.
M. L.- Y en la célula, ¿qué máquinas se utilizan para copiar la información?
R. A-G.- Se llaman polimerasas, y a diferencia de los cabezales ópticos de un grabador de DVD, son pequeños robots del tamaño del nanómetro que manipulan las hebras del ADN pegándose a ellas, algunos de ellos abriéndolas como una cremallera, y las replican según las van recorriendo. El ARN es otra molécula capaz de almacenar información de la misma forma en que lo hace el ADN, por lo tanto nosotros nos preguntamos: ¿Cuál es la ventaja tecnológica del ADN como disco duro en las células frente al ARN? A partir de medidas de la elasticidad de cada molécula de forma individual hemos podido concluir que el proceso de manipulación (apertura y lectura) de la doble hebra del ARN por motores biomoleculares tendría que ser muy diferente. Es muy probable que la eficiencia energética en la nano-escala sea una de las razones principales por las que la naturaleza ha optado por el ADN en la línea evolutiva, aunque ésta es una discusión que necesita de más enfoques para poder ser interpretada correctamente.
Mónica Luna es investigadora en Nanociencia y Nanotecnología en el Instituto de Microelectrónica de Madrid (CNM-CSIC).
Fuente: EL MUNDO.es




















0 comentarios:
Publicar un comentario